-
[R 데이터 분석] 계층적 군집분석머신러닝 with R 2019. 8. 31. 15:13
계층적 군집분석(Hierarchical Clustering)이란?
계층적 트리 모형을 이용해 개별 개체들을 순차적, 계층적으로 유사한 개체 내지 그룹과 통합하여 군집화를 수행하는 알고리즘이다. K-평균 군집과 달리 군집 수를 사전에 정의하지 않고, 학습 이후 군집수를 선택한다.
개체들이 결합되는 순서는 덴드로그램(Dendrogram)으로 시각화할 수 있어, 덴드로그램을 만들고 적절하게 트리를 잘라 군집을 나눌 수 있다.

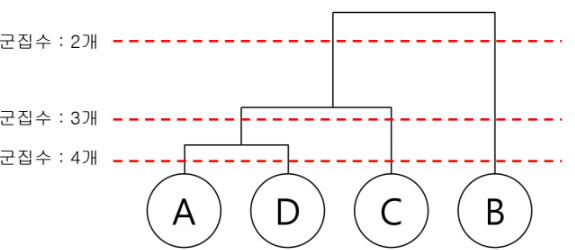
Dendrogram과 cutree로 군집화 한 결과 어떻게 만들 수 있을까?
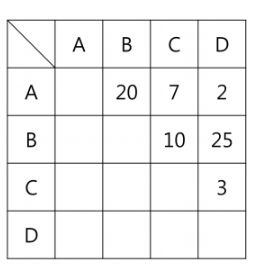
2) 가장 가까운 집단 찾기
- 거리는 수가 작을 수록 가깝고 클수록 멀다. -> A와 D의 거리가 2로 가장 가깝고, B와 D의 거리가 가장 멈
- 가장 가까운 집단을 묶는다. -> A와 D를 하나로 묶어줌.
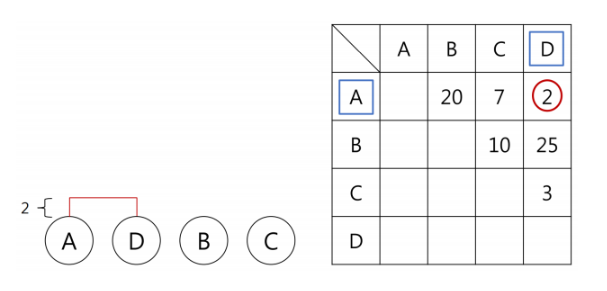
- 그렇다면 나머지 값들과의 거리는?? 어떻게 할 것인가?
3) 가장 가까운 개체와 군집 간의 거리
- 단일 연결
- single (최단연결) : 가장 가까운 개체끼리 연결
- complete (최장연결): 가장 먼 개체끼리 연결 (보수적으로)
- average (평균연결) : 모든 점들을 모두 연결하여 평균 계산함. 속도가 느려질 수 있지만 이상치에 덜 민감할 수 있다.
- centroid (중심연결): 군집의 중심을 잡아 거리를 계산. 평균보다 계산 양이 적어져 더 빠를 수 있음.

- Ward : 거리 행렬을 구할 때, 오차제곱의 증분을 두군집 사이의 거리로 측정
- K-means 에서 군집을 묶을 때 사용됨
- 단일 연결보다 이상치에 덜 민감함
--> 최단 연결을 사용했을 때 나머지 프로세스 진행

AD 그룹과 B의 거리는 20, AD 그룹과 C의 거리는 3 -> C 선택 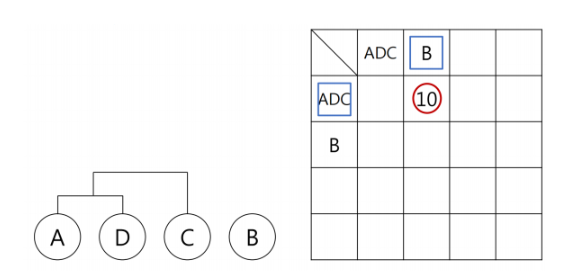
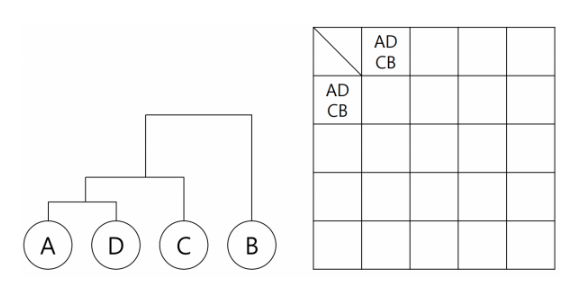
4) 군집수 선택
계층적 군집분석의 장단점
장점
- 구현이 간단하고 이해하기 쉬움
- 나무형 그림을 통해 군집화 과정을 볼 수 있음.
단점
- 하나의 군집이 형성되면 그 군집에 속한 개체들은 항상 같은 군집에 속함 -> 수정하지 못함
- 자료가 많은 경우 거리 비교 횟수가 늘어남 -> 대용량에 적합 X
예제 코드
Data: wine (in HDclassif pacakage)
#계층적 군집분석
if (require("HDclassif") == F) install.packages("HDclassif") #for sample data
if (require("NbClust") ==F) install.packages("NbClust") #for cluster profileling
if (require("cluster") == F) install.packages("cluster") #for sample data
if (require("sparcl") == F) install.packages("sparcl") #for visualization
if (require("compareGroups") == F) install.packages("compareGroups")
library(dplyr)
library(cluster)
library(compareGroups)
library(NbClust)
library(sparcl)
library(HDclassif)
#load data
data(wine)
str(wine)
names(wine) <- c("Class", "Alcohol", "MalicAcid", "Ash", "Alk_ash", "magnesium",
"T_phenols", "Flavanoids", "Non_flav", "Proantho", "C_Intensity",
"Hue", "OD280_315", "Proline")
wine_scale <- scale(wine[,-1]) #class 변수 제외
wine_scale <- as.data.frame(wine_scale) #scale 함수를 사용한 경우 데이터 프레임 형태로 바뀜
str(wine_scale)
#몇 개의 군집을 만들까?
numCoplte <- NbClust(wine_scale, distance = "euclidean", min.nc = 2, max.nc = 6, method = "complete", index = "all")
#index는 23개의 지표를 제공해주는데, 몇 개로 만드는 게 최적인가를 제공. all 은 클러스터 수를 제안을 받겠다는 의미
numCoplte$Best.nc
dis <- dist(wine_scale, method = "euclidean")
hc_1 <- hclust(dis, method = "complete") #계층적 군집 만들기 (최장 연결로)
str(dis)
par(mfrow = c(1,1))
plot(hc_1, hang = -1, lables = F, main = "Complete-Lingage")
com3_1 <- cutree(hc_1, 3) #3개의 군집으로 잘라라
ColorDendrogram(hc_1, y = com3_1, main = "Complete", branchlength = 20)
table(com3_1)
table(com3_1, wine$Class)
##ward 방식으로 만들기
numward <- NbClust(wine_scale, distance = "euclidean", min.nc = 2, max.nc = 6, method = "ward.D2", index = "all")
numward$Best.nc
hc_2 <- hclust(dis, method = "ward.D2")
par(mfrow = c(1,1))
plot(hc_2, hang = -1, labels = F, main = "ward's-Lingage")
ward3_1 <- cutree(hc_2, 3)
table(ward3_1, wine$Class)
#군집 간 비교
table(com3_1, ward3_1)
#군집 별 평균 비교
round(aggregate(wine[,-1], list(com3_1), mean),2)
round(aggregate(wine[,-1], list(ward3_1), mean),2)
wine$cluster <- com3_1
group <- compareGroups(cluster ~. , data = wine[,-1])
cluster_info <- createTable(group)
export2csv(cluster_info, file = "wine_cluster_compare.csv") #위 테이블을 파일로 저장[참고]
실무에 써먹는 머신러닝 with R
반응형'머신러닝 with R' 카테고리의 다른 글
[R 데이터 분석] 밀도기반 군집분석 (DBSCAN, Density-Based Spatial Clustering of Applications with Noise) (0) 2019.09.06 [R 데이터분석] k - 평균(k-means) 군집 분석 (0) 2019.09.02 [R 데이터 분석] 군집분석의 이해 :: 군집을 묶는 기준 '거리' / 군집분석의 평가 (0) 2019.08.30 R과 기초통계 - 좋은 회귀 모형? 회귀분석의 확장 LASSO (0) 2019.08.24 R 기초 통계/ 로지스틱 회귀 분석 (Logistic Regression)이란? (0) 2019.08.23